Übersetzung aus Cayman Chemical Currents: Autophagozytose und der Ubiquitin-Proteasom-Signalweg.
von Paul Domanski
Für den geregelten Ablauf der zellulären Funktionen sind wenige Faktoren so entscheidend wie das Gleichgewicht zwischen Aufbau bzw. Synthese und dem Abbau cytoplasmatischer Organellen und Proteine. Die Zelle regelt diese Balance hierbei primär über zwei Mechanismen: 1) Dem Ubiquitin-Proteasom-System und 2) Autophagozytose (‘Selbstfressen’), bei der zelluläre Komponenten dem lysosomalen Abbau zugeführt werden. Während das Ubiquitin-Proteasom-System primär kurzlebige und fehlgefaltete Proteine abbaut, wird die Degradation cytoplasmatischer Komponenten, beschädigter Organellen sowie langlebiger und aggregierter Proteine mittels Autophagozytose durchgeführt. Dieses zelluläre ‘Recycling’ System ist hochgradig reguliert und ermöglich somit die effiziente Rückführung und Regeneration der Zellbausteine.
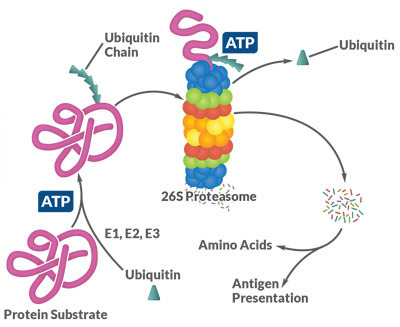
Abbildung 1. Die Zielproteine des Ubiquitin-Proteasom-Systems werden mittels eines dreischrittigen, ATP-abhängigen Mechanismus durch E1, E2 und E3 ubiquitiniert. Im Proteasom werden die Proteine anschließend entfaltet und zu kurzen Peptidketten abgebaut, die dann im Rahmen der Antigenpräsentation über MHC I verwendet werden können oder aber in die einzelnen Aminosäuren hydrolysiert werden.
Ubiquitinierung, eine der häufigsten posttranslationalen Modifikationen (PTM) in der Zelle, ist der Mechanismus durch den die Mehrzahl der kurzlebigen Proteine für Degradation und Abbau markiert werden. Es handelt sich hierbei um ein hochspezifisches System, bei dem Ubiquitin, ein mit 76 Aminosäuren eher kleines Protein, über eine Amidbindung mit einen Epsilon-Amin eines Lysins des Zielproteins verknüpft wird. Dies geschieht mittels eines dreischrittigen enzymatischen Prozesses.
Im Rahmen der hierarchischen Ubiquitinierungskaskade kann E1 dutzende E2 Proteine binden, welche wiederum die Möglichkeit haben mit hunderten E3 Proteinen zu assoziieren. Zudem können Proteine monoubiquitiniert, multiubiquitiniert und polyubiquitiniert werden. Dies erhöht die Variabilität des Abbausystems, da verschiedene Ubiquitinierungsmuster über die Bindung zu den mehr als 20 bekannten spezifischen Ubiquitinbindedomänen unterschiedliche Signalwege aktivieren können. Ähnlich wie die Phosphorylierung ist auch die Ubiquitinierung reversibel und ∼85 Deubiquitinasen (DUBs) wurden beschrieben. Die Mehrheit der ubiquitinierten Proteine wird, wie zuvor erläutert, dem Abbau zugeführt, allerdings kann die Modifikation mit Ubiquitin bei einigen Proteinen auch als Transportsignal wirken oder Protein-Protein-Interaktionen vermitteln (Abbildung 1).1-3
Ubiquitinierung ist bei diversen Zellprozessen und Fehlfunktionen involviert, hierzu gehören unter anderem die Immunantwort, DNA-Reparatur, Signaltransduktion, Krebserkrankungen, Neuropathien und viele mehr. Hieraus ergibt sich natürlich, dass Fehler im Ubiquitinierungsweg weitgehende und vielfältige Auswirkungen in der betroffenen Zelle haben und somit ursächlich an der Entstehung einer Vielzahl von neurodegenerativen, kardiovasculären und Tumorerkrankungen beteiligt sind.4,5 Im Rahmen der großen chemischen und technologischen Fortschritte (z.B. Massenspektrometrie) der letzten zwei Jahrzehnte werden daher nun immer mehr Wirkstoffe entwickelt, die in das Ubiquitin-Proteasom-System eingreifen.
Cayman Chemical bietet Ihnen ein breites Angebot an Produkten für die Erforschung der Rolle und Funktion des Ubiquitins und des Proteasoms. Ein Beispiel sind diverse Inhibitoren und Agonisten der Ubiquitinierung, die die gezielte Analyse einzelner Prozesse oder Enzyme im Ubiquitin-Proteasom-System ermöglichen. Zusätzlich bietet Cayman Ihnen eine Reihe von Antikörpern, unter anderem FK1 und FK2, mit denen Sie spezifische Ubiquitinierungsmuster auf den Zielproteinen unterscheiden können (Monoubiquitinierung, Multiubiquitinierung und Polyubiquitinierung). Cayman Chemical hat vor kurzem auch Kits für den Ubiquitinierungsnachweis, die Quantifizierung von Ubiquitin-bindenden Proteinen oder der Deubiquitinaseaktivität zum Portfolio hinzugefügt, die Wissenschaftlern als entscheidendes Werkzeug bei der weiteren Erforschung des Systems zur Seite stehen.
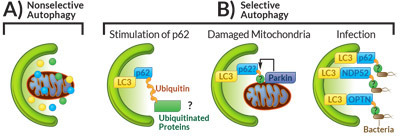
Abbildung 2. Nichtselektive und selektive Autophagozytose (A) Nichtselektive Autophagozytose wird unter Hungerstress aktiviert und vermittelt den Abbau cytoplasmatischer Komponenten in verwertbare kleinere Bausteine. (B) Stimuli wie beispielsweise die Aktivierung von p62, die Beschädigung von Mitochondrien oder Infektionen können die Aktivierung der selektiven Autophagozytose auslösen.
Autophagozytose ist ein evolutionär hochkonservierter zellulärer Abbauprozess, bei dem Zellkomponenten wie beispielsweise Organellen mittels der lysosomalen Maschinerie abgebaut werden. Folglich ist Autophagozytose ein normaler Teilprozess von Zellwachstum, -entwicklung, -differenzierung und -homöostase. Autophagozytose ermöglicht der Zelle ein diffiziles Gleichgewicht der Synthese, des Abbaus und des Recyclings zellulärer Komponenten beizubehalten. In niederen Organismen reguliert es das metabolische Gleichgewicht bei schwankender Nährstoffversorgung.6 Während ursprünglich davon ausgegangen wurde, dass es sich bei Autophagozytose um einen eher ‘kruden’ und wenig regulierten Prozess zur Zerstörung von Zellkomponenten handelt, wurde im Rahmen neuerer Erkenntnisse offensichtlich, dass Autophagozytose auch ‘selektiv’ abläuft. In höheren Organismen eliminiert dieses System aggregierte Proteine, beschädigte Organellen wie zu Beispiel Mitochondrien (Mitophagie) oder Peroxisomen (Pexophagie) und beseitigt zudem mikrobielle Pathogene (Xenophagie) (Abbildung 2).7,8 Zwar läuft Autophagozytose zumindest auf einem basalen Niveau dauerhaft in jeder Zelle ab, diese Aktivität kann aber in Folge externer und interner Stimuli dramatisch ansteigen. Diverse Signaltransduktionswege vermitteln diese stringente Regulation, allerdings sind die genauen Mechanismen dahinter momentan häufig nur unvollständig untersucht. Eine ganze Reihe von beteiligten Proteinen sind aber bekannt, unter anderem mTOR, p62, Beclin 1 und PINK1 (Abbildung 3).8 Da Autophagozytose genau wie Ubiquitinierung in konstanter Balance mit der Neusynthese von Proteinen und Organellen ablaufen muss, ist auch hierbei zu erwarten, dass Fehlregulationen des Systems weitreichende, pathologische Folgen für den Organismus haben. Tatsächlich konnte gezeigt werden, dass Knockout-Mäuse, denen ein für Autophagozytose entscheidendes Gen fehlt, innerhalb von Stunden nach der Geburt versterben.9 Es ist außerdem bekannt, dass Störungen in der Autophagozytose eine Rolle spielen bei Krebs, neurodegenerativen Erkrankungen (sowohl bei der Huntington und der Alzheimer Krankheit zeigt sich eine erhöhte Akkumulation von autophagischen Vakuolen), lysosomalen Speicherkrankheiten, Infektionskrankheiten und der Reparatur von DNA-Schäden (über HDACs). Es wird außerdem vermutet, dass Autophagozytose-Fehlfunktionen an Alterung und Zelltod beteiligt sind.10,11
Autophagozytose ist primär durch die Präsenz eines einzigartigen Organells gekennzeichnet, dem Autophagosom. Das Autophagosom ist eine mit einer Doppelmembran umgebende Vakuole mit einem Durchmesser von 300-900 nm. Es entsteht aus dem sogenanntem Phagophor, welches sich ausdehnt und hierbei cytoplasmatische Komponenten einschließt. Diese Phagosomen fusionieren anschließend mit dem Lysosom und bilden somit das aktive Autolysosom.6
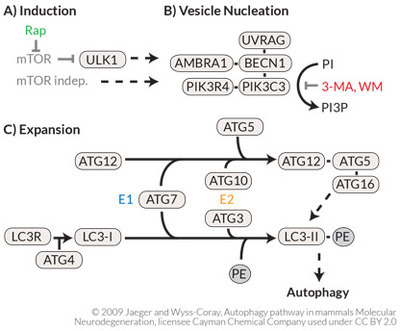
Abbildung 3. Der Autophagozytoseweg in Säugetieren (A) Die prototypische Induktion der Autophagozytose durch mTOR (inhibiert durch Rapamycin) führt zur (B) Nukleation/Erweiterung der autophagosomalem Membran mit Hilfe eines Beclin 1 und PI3K- enthaltenden Proteinkomplexes (inhibiert durch 3-Methyladenin (3-MA) und Wortmannin). (C) Die Expansion der Membran erfolgt durch einen der Ubiquitinierung ähnlichen Mechanismus, bei dem ATG7 und ATG3 LC3-I zu LC3-II umwandeln, welches wiederum an der autophagosomalen Membran akkumuliert.
In den späten 1990ern führten Arbeiten an Hefen zur Entdeckung von über 20 Autophagozytose-beteiligte-Genen (ATGs). Weitere Forschung führte schließlich zur Beschreibung der Säugetier-Homologe dieser Gene und der zugehörigen Proteine.12,13 Eine schematische Darstellung der Signalkaskade sowie der bekannten Inhibitoren und Aktivatoren ist in Abbildung 3 gezeigt. Unter nährstoffreichen Bedingungen agiert die Proteinkinase mTOR als primärer negativer Regulator der Autophagozytose. Unter Hungerbedingungen wird allerdings mTOR (das Rapamycin Zielprotein im Säuger) deaktiviert und somit ULK (das Säugerequivalent des Hefeproteins ATG1) aktiviert, welches anschließend in das endoplasmatische Retikulum transloziert und dort die Autophagozytose-Kaskade initiiert. Die Formation des Phagosoms wird abgeschlossen, indem ATG8 (in Säugetieren die leichte Kette 3 des Mikrotubuli-assozierten Proteins (MAP-LC3 oder LC3-I)) C-terminal durch ATG4 gespalten und Phosphatidylethanolamin (PE) an ATG8 gekoppelt wird. Dieses LC3-Phospholipid (LC3-II) bindet anschließend kovalent an das Phagophor und ist essentiell für die Bildung des Phagosoms. Aus diesem Grund kann LC3-II als hochspezifischer Marker für das Autophagosom verwendet werden. Genau wie die Ubiquitinierung ist auch dieser Prozess reversibel, und diese Delipidisierung wird ebenfalls über ATG4 vermittelt.
Die Autophagozytoseaktivität in Zellen kann mittels vieler verschiedener Methoden quantifiziert werden. Zu den ersten erfolgreichen Ansätzen gehörte das Anfärben der Autophagosomen mit acidotropen Farbstoffen, wie Monodansylcadaverin (MDC) (Cayman’s Autophagy/ Cytotoxicity Dual Staining Kit) sowie Transmissions-Elektronenmikroskopie (TEM).14
Interessanterweise verursacht die PE-Modifikation von LC3-I eine Veränderung der Elektrophoresemobilität, welche die genaue Analyse der LC3-I und LC3-II-Level per Western Blot ermöglicht. In Kombination mit lysosomalen Inhibitoren wie Bafilomycin A1 kann mit dieser Technik die Akkumulation von LC3-II im Autophagolysosom gemessen werden. Dies wiederum erlaubt die Bestimmung des ‘Autophagischen Flusses’, das heißt der Rate mit der Proteine und Zellmaterial im Autophagosom prozessiert werden. Dies ist das Prinzip hinter dem Cayman Autophlux Kit, welches Wissenschaftlern durch die Analyse von LC3-II und p62 (einem bekannten LC3-bindenden Protein) unter Gabe von Bafilomycin A1 die Bestimmung des Autophagischen Flusses unter diversen experimentellen Bedingungen ermöglicht.14-17
Zusätzlich bietet Cayman eine Reihe von chemischen Modulatoren der Autophagozytose sowie ein LC3 Interact Kit an, dass zur Identifikation von Autophagozytose-regulierenden Proteinen eingesetzt werden kann. Das Ubiquitin-Proteasom-System und Autophagozytose sind von entscheidender Bedeutung für das diffizile Gleichgewicht der Produktion und Degradation von Proteinen und Organellen in allen Zellen. Heute ist bekannt, dass diese Abbauwege auch interagieren können und, dass Autophagozytose unter extremen Stress auch ubiquitinierte Proteine abbaut. Obwohl vieles schon entschlüsselt wurde, ist noch einiges an Forschung notwendig. Forscher weltweit arbeiten daran neue Biomarker zu identifizieren, um die Funktionen dieser Systeme genauer zu verstehen und innovative Therapiemöglichkeiten für die assoziierten Krankheiten zu entwickeln.
Learn more about Cayman Chemical